大家好,本周为大家分享一篇发表在Analytical Chemistry上的文章,High-Sensitivity Proteome-Scale Searches for Crosslinked Peptides Using CRIMP 2.0,该文章的通讯作者是卡尔加里大学的David C. Schriemer教授。
交联质谱(XL-MS)是一种在蛋白质空间中生成点对点的距离测量值的有价值的技术。然而,基于细胞的XL-MS实验需要高效的软件来灵敏及错误率可控地检测交联肽。已经有许多算法通过过滤策略,在进行交联搜索之前达到缩减数据库的大小的效果,但也有人担心使用这些策略可能会降低灵敏度。
本文提出了一种新的评分方法,使用快速预搜索方法和受计算机视觉算法启发的概念来解析来自其他冲突反应产物的交联。对几个精选的交联数据集的搜索显示了很高的交联检测率,即使是最复杂的蛋白质组水平的搜索(使用不可断裂或可断裂的交联剂)也可以在传统的台式计算机上高效地完成。通过在评分方程中包含组成项,蛋白质−蛋白相互作用的检测增加了两倍。该组合功能可在软件Mass Spec Studio中作为CRIMP 2.0提供。
CRIMP 2.0 集成了改进的库缩减引擎和新的评分算法,可解决所有类别命中(例如游离肽、单链和交联)的谱图冲突。文章中修订后的误差估计方法考虑了在其他搜索工具中大多被忽略的跨类别的谱图冲突,并支持检测蛋白质-蛋白质相互作用的新方法。本文证明了库缩减策略确实可以提供高灵敏度,并支持不可断裂和可断裂实验类型的全蛋白质组分析,并且只需使用很少的计算资源。
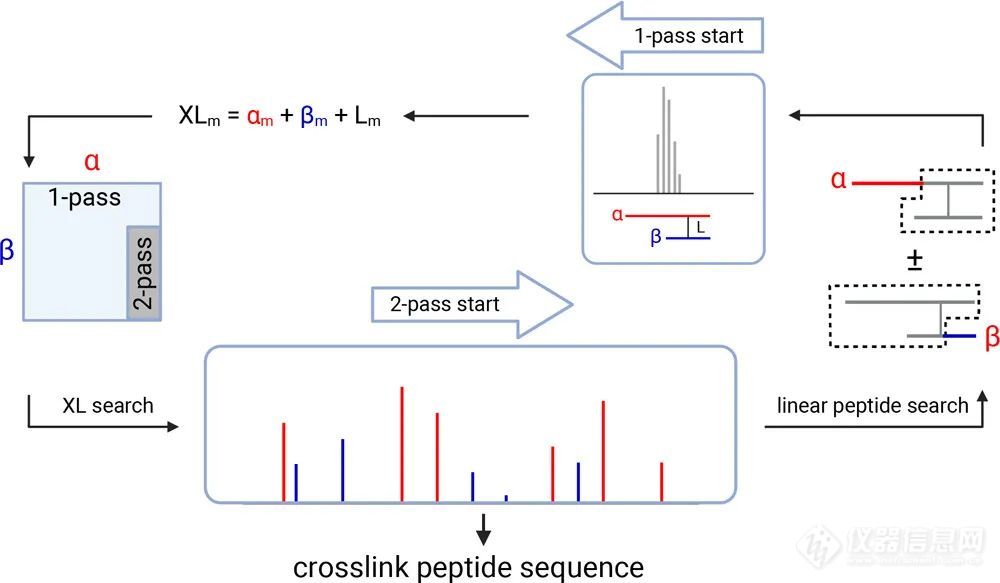
图1 概述了搜索MS2数据集以寻找肽交联证据的典型方法的示意图。单通道方法从假定的交联肽的前体质量开始,并通过一个涉及α肽质量、β肽质量和交联剂质量的简单的三项加和来限制数据库搜索。然后,在MS2谱图中搜索组合。双通道方法由MS2谱图开始并结束:首先,在MS2数据中发现了候选α和β肽,此时,前体质量才用于限制组合,以便对MS2数据进行更详尽的搜索。
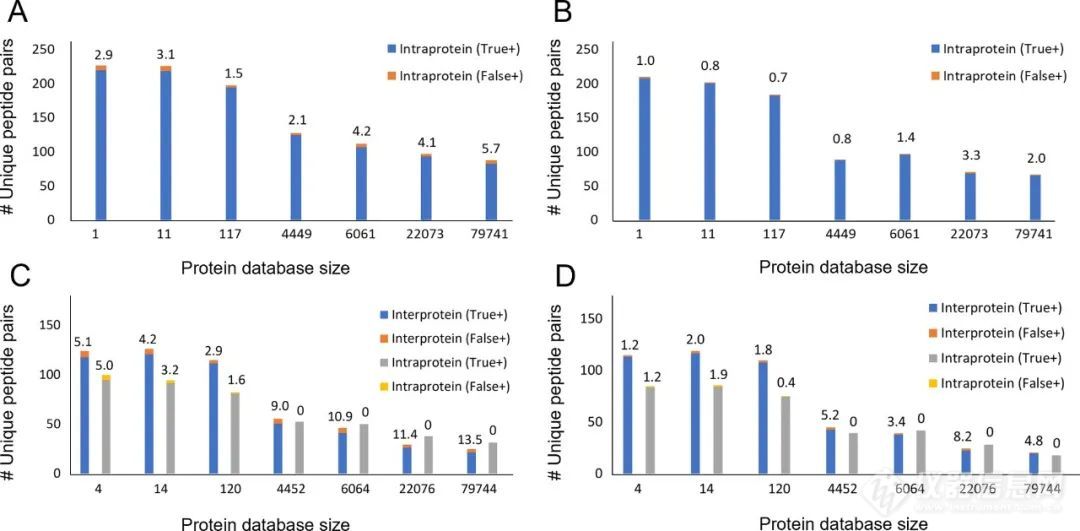
图2 使用Beveridge等人研究的DSS交联剂的复制数据集测试交联灵敏度。以(A)为5%和(B)为1%的计算FDR值对Cas9数据库进行了分析。分段Cas9数据库的结果在(C)为5%和(D)为1% FDR,显示为蛋白内和蛋白间搜索结果。使用多个添加的数据库显示诱饵数据库的效果,并注意到蛋白质的复杂性。真实的%FDR展示为标注数字。
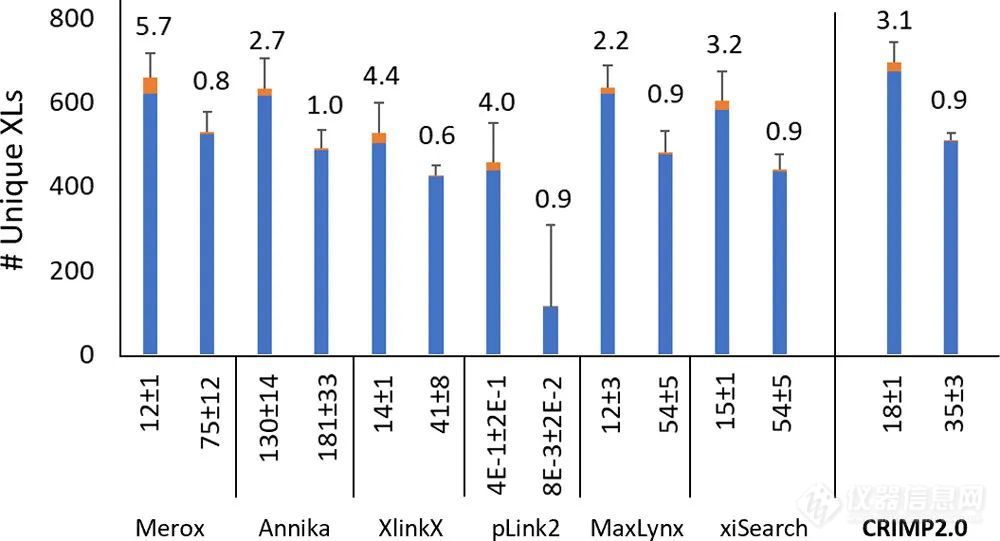
图3 使用 DSSO 作为交联试剂和阶梯式HCD MS2 方法进行数据采集的平均交联肽段数目。上述算法的所有结果均来自添加了 CRIMP 2.0 的 Matzinger 等人,预期 FDR 为 1%(成对的左条),并使用分数后截止显示校正结果以达到实验验证的 FDR 1%(成对的右条)。
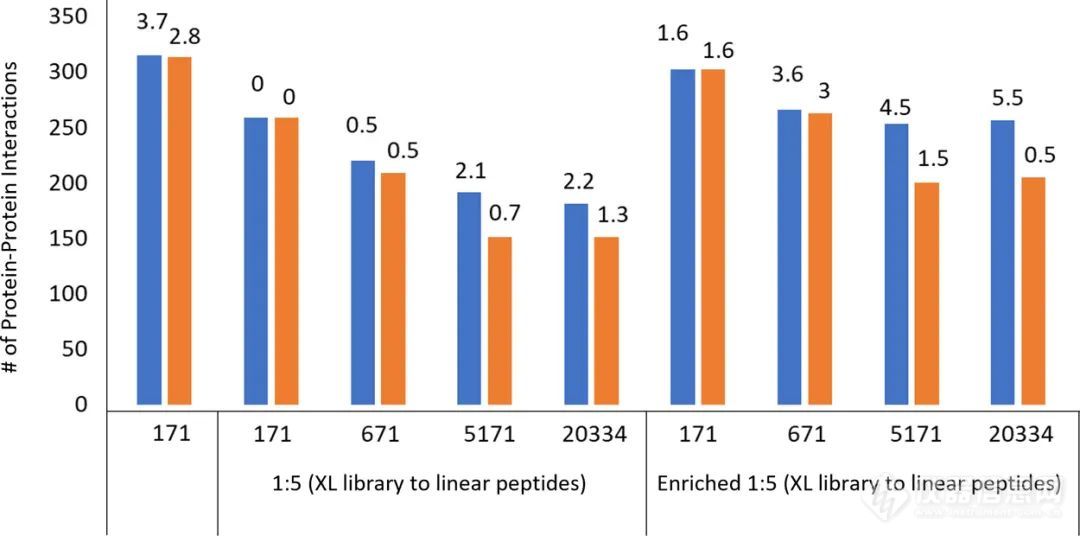
图4 合成肽基准数据集2中检测到的 PPI 数量,来自Matzinger 等人的研究。
蓝色条表示以估计的 5% FDR 进行的搜索,橙色条表示以估计的 1% FDR 进行的搜索。检索基准中的所有三组数据,并在搜索数据库中使用指示数量的蛋白质来探索诱饵的影响。真实的%FDR展示为标注数字。
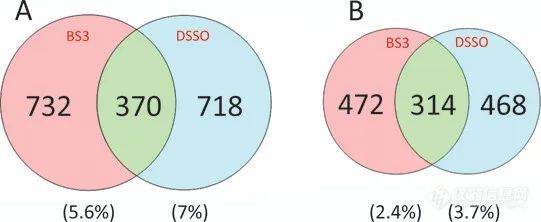
图4 使用两种交联试剂交联大肠杆菌蛋白质组的 PPI 搜索结果。大肠杆菌蛋白质组使用两种交联试剂。(A)以目标5%FDR进行的搜索和(B)以目标1%FDR进行的搜索。结果基于Lenz等人研究中建立的近似PPI数据库,使用成分知情PPI评分方法。图底部的百分比显示了基于库组成的计算出的 FDR 值。
本文的结果表明,双通道数据库简化方法可以返回复杂样品中交联组成的灵敏测量。控制数据库限制的程度允许用户调整搜索速度以满足实验的需要,而不会引起对极大的灵敏度损失,因为对搜索参数的依赖性是适度且可预测的。通过对关键搜索词(如N,Eα和Eβ)进行细微的调整,即使是人类蛋白质组和密集的数小时LC - MS / MS运行也可以在一天或更短的时间内在一台台式计算机上进行处理,例如本研究中使用的那样。对于高度复杂的系统,蛮力穷举方法可能被证明不如双通道方法敏感。数据库的不必要扩展可能会产生嘈杂的搜索,就像蛋白质组学搜索使用过多的变量修改进行参数化时所做的那样。CRIMP允许对可裂解和不可裂解的交联剂进行强健的搜索,而不可裂解试剂在原位应用中应得到更多关注。这些试剂更容易合成,并且在这种规模上显然是互补的。此外,这些试剂产生跨肽片段离子,这可能是验证命中值的必要条件,特别是在探索相互作用由翻译后修饰定义的高度复杂状态时。总而言之,本文提出的 CRIMP 2.0 提供了此类活动所需的灵敏度和搜索速度。
撰稿: 聂旻涵编辑: 李惠琳
原文: High-Sensitivity Proteome-Scale Searches for Crosslinked Peptides Using CRIMP 2.0
参考文献
1. Crowder DA, Sarpe V, Amaral BC, Brodie NI, Michael ARM, Schriemer DC. High-Sensitivity Proteome-Scale Searches for Crosslinked Peptides Using CRIMP 2.0. Anal Chem. 2023;95(15):6425-6432. doi:10.1021/acs.analchem.3c00329
